이 포스트는 Do it! 정직하게 코딩하며 배우는 딥러닝 입문 pp.117~154를 참고하였습니다.
검증 세트를 나누고 전처리 과정 배우기
먼저, 이전 포스트에서 마지막에 사용했던 SGDClassifier 클래스를 사용하여 모델을 훈련하고 평가해보겠습니다.
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import train_test_split
from sklearn.linear_model import SGDClassifier
cancer = load_breast_cancer()
x = cancer.data
y = cancer.target
x_train_all, x_test, y_train_all, y_test = train_test_split(x, y, stratify=y, test_size=0.2, random_state=42)
sgd = SGDClassifier(loss='log', random_state=42)
sgd.fit(x_train_all, y_train_all)
sgd.score(x_test, y_test)
위 훈련 결과, 테스트 세트의 정확도는 약 83%입니다. 이 성능이 맘에 들지 않는다면, 다른 손실 함수를 사용해도 됩니다. 이렇게 손실 함수, loss와 같은 매개변수 값은 가중치나 절편처럼 스스로 학습하는 것이 아닌, 사용자가 직접 정해야 하는 파라미터입니다. 이런 파라미터를 하이퍼파라미터(hyperparameter)라고 합니다. 이런 하이퍼파라미터를 바꾸는 작업을 '모델을 튜닝한다'고 합니다.
여기서 문제가 하나 있는데, 위의 결과가 맘에 들지 않아서 하이퍼파라미터를 바꾼다면 그건 올바른 훈련 방법이 아닙니다. 위의 결과는 테스트 세트의 결과인데, 이 결과를 보고 모델을 튜닝하면 테스트 세트의 결과만 좋게 나오는 모델이 나오게 되며, 이는 이전에 말했듯이 기출문제의 답만 외우는 것과 같습니다. 우리가 원하는건 테스트 성능만 좋은 모델이 아니라 실전 성능이 좋은 모델입니다.
검증 세트 준비
따라서, 모델을 튜닝 할 때는 테스트 세트를 사용하지 않아야 합니다. 테스트 세트는 모델 튜닝을 마치고 실전에 사용하기 전 딱 한번만 사용하는 것을 권장합니다. 즉, 모델 튜닝에 필요한 세트는 따로 준비해야 하는데, 이를 검증 세트(validatoin set)라고 하며, 훈련 세트의 일부를 사용하여 만듭니다. 이번에는 훈련 세트, 검증 세트, 테스트 세트의 비율을 60%, 20%, 20%로 할당하여 모델을 훈련해보겠습니다.
데이터 세트 준비하기
이번에도 위스콘신 유방암 데이터를 사용할 것입니다.
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import train_test_split
cancer = load_breast_cancer()
x = cancer.data
y = cancer.target
x_train_all, x_test, y_train_all, y_test = train_test_split(x, y, stratify=y, test_size=0.2, random_state=42)
검증 세트 분할하기
방금 훈련, 검증, 테스트 세트의 비율을 6:2:2정도로 분할한다고 했는데, 실제 분할 작업은 처음부터 이 비율로 나누는 것이 아니라 먼저 전체 데이터 세트를 8:2로 나누고, 훈련 세트를 다시 8:2로 나누어 검증 세트를 만듭니다. 방금 전체 데이터 세트를 8:2로 나누었으니 이제 훈련 세트를 다시 8:2로 나누어서 검증 세트를 만듭니다.
x_train, x_val, y_train, y_val = train_test_split(x_train_all, y_train_all, stratify=y_train_all, test_size=0.2, random_state=42)
print(len(x_train), len(x_val))
검증 세트로 모델 평가하기
sgd = SGDClassifier(loss='log', random_state=42)
sgd.fit(x_train, y_train)
sgd.score(x_val, y_val)
훈련 결과, 정확도가 약 83%에서 약 69%로 줄었습니다. 이는 훈련 데이터의 수가 많이 줄었기 때문입니다. 이렇게 데이터가 적은 경우에는 교차 검증(cross validation)이라는 방법을 사용하기도 합니다. 이에 대한 내용은 나중에 설명하겠습 니다.
데이터 전처리와 특성의 스케일
사이킷런 같은 머신러닝 패키지에 준비된 데이터는 대부분 실습을 위한 것이므로 잘 가공되어 있으나, 실전에서 수집된 데이터는 그렇지 않습니다. 누락된 값이 있을 수도 있고, 데이터의 형태가 균일하지 않을 수도 있습니다. 이런 데이터를 가공하는 것을 데이터 전처리(data preprocessing)이라고 합니다.
특성의 스케일
이 책에서는 누락된 데이터는 다루지 않는데, 이런 경우의 데이터 전처리 방법을 간단하게 설명드리자면 누락된 데이터가 있는 샘플을 아예 없애거나, 평균 값으로 대체하거나, 가장 많이 나타나는 값으로 대체하는 등 몇 가지의 방법이 있습니다.
이미 정리된 데이터도 전처리를 해야하는 경우가 있긴 한데, 이는 특성의 스케일(scale)이 다른 경우에 해당합니다. 특성의 스케일이란, 어떤 특성이 가진 값의 범위를 의미합니다. 예를 들어, 어떤 과일을 분류하는데, 특성 중에 당도와 무게가 있다고 가정하겠습니다. 과일의 당도는 보통 20을 넘지 않고, 무게는 100g정도부터 1000g이상까지 나오는 과일도 있을 것입니다. 이런 경우, 두 특성의 스케일 차이가 크다고 합니다.
어떤 알고리즘들은 이렇게 특성의 스케일에 민감하며, 경사 하강법도 스케일에 민감한 알고리즘 중 하나이기 때문에 특성의 스케일을 조정해야 합니다.
스케일을 조정하지 않고 모델을 훈련하는 경우
이제 특성의 스케일이 서로 다른 데이터를 이용하여 훈련했을 때의 결과를 보겠습니다.
훈련 데이터 준비하고 스케일 비교하기
이전 포스트에서 위스콘신 유방암 데이터에서 몇 개의 특성이 매우 큰 것을 봤습니다. 이번엔 그 데이터들을 사용할 것이며, 단일층 신경망 모델을 만들어 훈련해볼 것입니다.
%matplotlib inline
import matplotlib.pyplot as plt
print(cancer.feature_names[[2, 3]])
plt.boxplot(x_train[:, 2:4])
plt.xlabel('feature')
plt.ylabel('value')
plt.show()

두 특성의 값을 보니, mean perimeter는 100~200사이의 값들이고, mean area는 200~2000사이의 값임을 알 수 있습니다. 이 두 값에 경사 하강법을 적용해봅니다. 기존에 사용했던 SingleLayer를 수정할 것입니다.
수정된 전체 코드는 다음과 같습니다.
import numpy as np
class SingleLayer:
def __init__(self, learning_rate=0.1):
self.w = None
self.b = None
self.losses = []
self.w_history = []
self.lr = learning_rate
def forpass(self, x):
z = np.sum(x*self.w) + self.b
return z
def backprop(self, x, err):
w_grad = -err*x
b_grad = -err*1
return w_grad, b_grad
def activation(self, z):
a = 1 / (1 + np.exp(-z))
return a
def fit(self, x, y, epochs=100):
self.w = np.ones(x.shape[1])
self.b = 0
self.w_history.append(self.w.copy())
np.random.seed(42)
for _ in range(epochs):
loss = 0
indexes = np.random.permutation(np.arange(len(x)))
for i in indexes:
z = self.forpass(x[i])
a = self.activation(z)
err = y[i] - a
w_grad, b_grad = self.backprop(x[i], err)
self.w -= self.lr*w_grad
self.b -= b_grad
self.w_history.append(self.w.copy())
a = np.clip(a, 1e-10, 1-1e-10)
loss += -(y[i]*np.log(a) + (1 - y[i])*np.log(1 - a))
self.losses.append(loss/len(y))
def predict(self, x):
z = [self.forpass(x_i) for x_i in x]
return np.array(z) > 0
def score(self, x, y):
return np.mean(self.predict(x) == y)
가중치를 기록할 변수와 학습률 파라미터 추가하기
def __init__(self, learning_rate=0.1):
self.w = None
self.b = None
self.losses = []
self.w_history = []
self.lr = learning_rate
SingleLayer 클래스에 가중치의 변화를 기록하기 위한 변수를 추가하고, 학습률이라는 파라미터도 추가합니다.
learning_rate는 하이퍼파라미터이며, 가중치가 업데이트 되는 크기를 조절합니다. 경사 하강법에서 기울기를 따라 내려갈 때에는, 조금씩 내려가야 합니다. 마치 산에서 눈을 감고 경사를 따라 골짜기로 내려가는 것을 생각하면 됩니다. 보폭을 크게 하면 낭떠러지로 떨어질 수도 있기 때문에, 조금씩 발걸음을 떼야 할 것입니다. 그렇다고 보폭이 너무 좁으면 내려가는데 한참 걸릴 것이니, 적당한 보폭을 유지하는 것이 좋을 것입니다.
경사 하강법에서는 보폭이 클 경우, 즉 학습률이 너무 큰 경우에는 손실 함수의 최저점을 제대로 찾지 못하거나, 기울기가 발산해버릴 수도 있습니다. 다음 그림에서 빨간색 화살표는 적절한 학습률 경우이고, 파란색 화살표는 너무 큰 학습률의 경우입니다.

가중치 기록하고 업데이트 양 조절하기
def fit(self, x, y, epochs=100):
self.w = np.ones(x.shape[1])
self.b = 0
self.w_history.append(self.w.copy())
np.random.seed(42)
for _ in range(epochs):
loss = 0
indexes = np.random.permutation(np.arange(len(x)))
for i in indexes:
z = self.forpass(x[i])
a = self.activation(z)
err = y[i] - a
w_grad, b_grad = self.backprop(x[i], err)
self.w -= self.lr*w_grad
self.b -= b_grad
self.w_history.append(self.w.copy())
a = np.clip(a, 1e-10, 1-1e-10)
loss += -(y[i]*np.log(a) + (1 - y[i])*np.log(1 - a))
self.losses.append(loss/len(y))이제 fit() 메서드에서 가중치들을 w_history에 기록합니다. numpy에서는 배열을 리스트에 추가하면 실제 값이 복사되는 것이 아니라 배열을 참조합니다. 따라서 원본이 바뀌지 않게 self.w의 값이 바뀌면 그 값을 복사하여 w_history에 추가합니다. 또, w_grad에 학습률 파라미터 self.lr를 곱해서 학습률을 조절합니다.
모델 훈련하고 평가하기
스케일을 조절하지 않은 상태에서 모델을 훈련하고 점수를 확인해보겠습니다.

점수가 약 91%를 정확도를 보여줍니다. 이번에는 스케일이 크게 차이났던 두 개의 가중치를 확인해보겠습니다.
w2 = []
w3 = []
for w in layer1.w_history:
w2.append(w[2])
w3.append(w[3])
plt.plot(w2, w3)
plt.plot(w2[-1], w3[-1], 'ro')
plt.xlabel('w[2]')
plt.ylabel('w[3]')
plt.show()
최종적으로 결정된 가중치는 빨간 점으로 나타내었습니다. 아까 데이터를 확인했을 때, mean area는 w[2]이고, mean perimeter는 w[3]이었으며, mean perimeter의 스케일이 더 컸습니다. 따라서 위의 그래프를 보면 w[3]이 학습 과정에서 큰 폭으로 진동하는 것을 볼 수 있습니다. 이 현상을 'w3에 대한 그래이디언트가 크기 때문에 w3축을 따라 가중치가 크게 흔들린다'라고 합니다. 이 현상을 해결하기 위해 스케일 조정이 필요합니다.
스케일을 조정해 모델을 훈련합니다
스케일을 조정하는 방법 중에서 신경망에서 자주 사용하는 스케일 조정 방법 중 하나는 표준화(standardization)입니다. 정규화(normalization)는 스케일을 0~1의 범위로 만드는 것입니다. 헷갈리지 마세요. 표준화는 특성값에서 평균을 빼고 표준 편차로 나눕니다. 그러면 평균이 0이고 분산이 1인 특성이 만들어집니다. 표준화 공식은 다음과 같습니다.
$z = \frac{x-\mu}{\sigma}$
numpy로 표준화 구현하기
numpy의 mean(), std() 함수를 사용하면 표준화를 쉽게 구할 수 있습니다. 매개변수 axis는 0일경우 행의 값이 커지는 방향으로(세로방향) 계산, 1일경우 열의 값이 커지는 방향으로(가로방향) 계산을 합니다.
train_mean = np.mean(x_train, axis=0)
train_std = np.std(x_train, axis=0)
x_train_scaled = (x_train - train_mean) / train_std
모델 훈련하기
이제 스케일을 조정한 데이터로 다시 트레이닝을 해보겠습니다.
layer2 = SingleLayer()
layer2.fit(x_train_scaled, y_train)
w2 = []
w3 = []
for w in layer2.w_history:
w2.append(w[2])
w3.append(w[3])
plt.plot(w2, w3)
plt.plot(w2[-1], w3[-1], 'ro')
plt.xlabel('w[2]')
plt.ylabel('w[3]')
plt.show()
모델 성능 평가하기
검증 세트를 사용하여 모델을 평가해봅니다.
layer2.score(x_val, y_val)
결과가 매우 좋지 않은데, 이 모델은 훈련 세트와 검증 세트의 스케일이 비슷할 것이라고 기대하기 때문입니다. 우리는 훈련 데이터만 전처리를 하였으므로, 검증 세트도 전처리 후 다시 성능을 확인해보겠습니다.
val_mean = np.mean(x_val, axis=0)
val_std = np.std(x_val, axis=0)
x_val_scaled = (x_val - val_mean) / val_std
layer2.score(x_val_scaled, y_val)
스케일을 조절할 때 주의해야 할 점을 알아봅니다
스케일을 조절할 때 주의해야 할 점은, 훈련 세트와 검증 세트는 같은 스케일로 조정되어야 한다는 것입니다. 방금 전에는 훈련 세트는 훈련 세트의 평균과 표준편차로, 검증 세트는 검증 세트의 평균과 표준편차를 사용하여 스케일을 조정하였습니다. 이런 경우에 데이터가 어떻게 나타나는지 확인해보겠습니다.
먼저, 원본 데이터의 산점도를 확인해봅니다.
plt.plot(x_train[:50, 0], x_train[:50, 1], 'bo')
plt.plot(x_val[:50, 0], x_val[:50, 1], 'ro')
plt.xlabel('feature 1')
plt.ylabel('feature 2')
plt.legend(['train set', 'val set'])
plt.show()
다음으로, 전처리된 데이터의 산점도를 확인해봅니다.
plt.plot(x_train_scaled[:50, 0], x_train_scaled[:50, 1], 'bo')
plt.plot(x_val_scaled[:50, 0], x_val_scaled[:50, 1], 'ro')
plt.xlabel('feature 1')
plt.ylabel('feature 2')
plt.legend(['train set', 'val set'])
plt.show()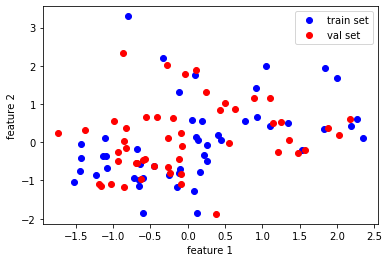
따로 놓고 보니 뭐가 다른지 잘 모르겠네요. 같이 놓고 확인해보겠습니다.

표시된 부분을 보면 훈련 세트와 검증 세트가 미세하게 다른 비율로 변환된 것을 알 수 있습니다. 즉, 원본 훈련 세트와 검증 세트의 점들 사이의 거리가 그대로 유지되지 않은 것입니다.
올바른 방법으로 검증 세트 전처리하기
앞서 언급했듯이, 올바른 데이터 전처리 방법은 훈련 세트와 검증 세트를 같은 비율로 전처리하는 것입니다. 훈련 세트의 평균과 표준 편차를 사용하여 검증 세트를 변환해야합니다.
x_val_scaled = (x_val - train_mean) / train_std
plt.plot(x_train_scaled[:50, 0], x_train_scaled[:50, 1], 'bo')
plt.plot(x_val_scaled[:50, 0], x_val_scaled[:50, 1], 'ro')
plt.xlabel('feature 1')
plt.ylabel('feature 2')
plt.legend(['train set', 'val set'])
plt.show()
이렇게 전처리를 제대로 한 데이터로 다시 모델의 성능을 평가 해도 기존과 같은 결과가 나오는데, 이는 데이터 세트가 별로 크지 않기 때문입니다. 검증 세트가 클 경우에는 차이가 날 수 있습니다.
과대 적합과 과소 적합을 알아보기
과대 적합이란 훈련 세트에서는 좋은 성능을 보이지만 검증 세트에서는 낮은 성능 보이는 것을 말합니다. 예를 들어 훈련 세트에서의 정확도가 99%이고 검증 세트의 정확도가 80%정도 같은 경우를 말합니다. 과소 적합은 훈련 세트에서도, 검증 세트에서도 성능이 낮은 경우를 말합니다.
다음과 같은 그래프를 학습 곡선(learning curve)라고 합니다.

첫 번째 그래프는 과대적합의 전형적인 형태입니다. 훈련 세트와 검증 세트의 성능의 간격이 크기 때문에 '분산이 크다(high variance)'라고도 합니다. 과대 적합의 주요 원인 중 하나는 훈련 세트에 충분히 다양한 패턴의 샘플이 포함되지 않은 경우입니다. 즉, 특정한 패턴만을 학습했기 때문에 학습했던 패턴을 별로 보이지 않는 샘플에 적용할 경우, 즉 일반화 성능은 떨어질 수 밖에 없습니다. 이런 경우에는 더 많은 훈련 샘플이 있으면 해결될 수도 있고, 모델이 훈련 세트에 집착하지 않게 가중치를 제한할 수도 있습니다. 이를 '모델의 복잡도를 낮춘다'라고도 합니다. 모델 복잡도에 대한 설명은 나중에 하겠습니다.
두 번째 그래프는 과소적합의 전형적인 형태입니다. 훈련 세트와 검증 세트에서 측정한 성능의 간격이 가까워지긴 하지만, 그 성능 자체가 낮기 때문에 '편향이 크다(high bias)' 라고도 합니다. 과소 적합은 모델이 충분히 복잡하지 않아 훈련 데이터에 있는 패턴을 모두 잡아내지 못하는 현상입니다. 이를 해결하는 방법은 복잡도가 더 높은 모델을 사용하거나 가중치의 제한을 완화하는 것입니다.
다음 그래프를 보겠습니다.
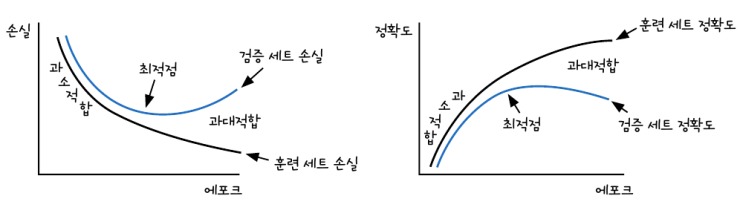
왼쪽 그래프를 보면, 에포크가 진행될 수록 훈련 세트와 검증 세트의 손실이 감소하지만, 어느 지점부터는 검증 세트의 손실이 다시 증가하고, 훈련 세트의 손실은 계속 감소하는 것을 볼 수 있습니다. 이 부분에서 과대적합이 시작되는 것입니다. 모델을 훈련할 때 가장 적절한 에포크 수는 모델이 과대적합되기 직전의 에포크입니다.
앞에서 과소적합된 모델은 '편향이 크다'라고 했고, 과대적합된 모델은 '분산이 크다'라고 했습니다. 편향과 분산은 트레이드-오프 관계에 있습니다. 따라서 우리는 분산이나 편향이 너무 커지지 않게 적절한 지점을 선택해야 합니다. 즉, 모델이 과대적합되지도, 과소적합되지도 않게 적절한 지점을 선택해야하는 것입니다.
이번에는 적절한 지점을 선택하는 것을 직접 구현해보겠습니다. 기존에 사용했던 SingleLayer 클래스를 약간 수정합니다.
전체 코드는 다음과 같습니다.
class SingleLayer:
def __init__(self, learning_rate=0.1):
self.w = None
self.b = None
self.losses = []
self.val_losses = []
self.w_history = []
self.lr = learning_rate
def forpass(self, x):
z = np.sum(x*self.w) + self.b
return z
def backprop(self, x, err):
w_grad = -err*x
b_grad = -err*1
return w_grad, b_grad
def activation(self, z):
a = 1 / (1 + np.exp(-z))
return a
def fit(self, x, y, epochs=100, x_val=None, y_val=None):
self.w = np.ones(x.shape[1])
self.b = 0
self.w_history.append(self.w.copy())
np.random.seed(42)
for _ in range(epochs):
loss = 0
indexes = np.random.permutation(np.arange(len(x)))
for i in indexes:
z = self.forpass(x[i])
a = self.activation(z)
err = y[i] - a
w_grad, b_grad = self.backprop(x[i], err)
self.w -= self.lr*w_grad
self.b -= b_grad
self.w_history.append(self.w.copy())
a = np.clip(a, 1e-10, 1-1e-10)
loss += -(y[i]*np.log(a) + (1 - y[i])*np.log(1 - a))
self.losses.append(loss/len(y))
self.update_val_loss(x_val, y_val)
def predict(self, x):
z = [self.forpass(x_i) for x_i in x]
return np.array(z) > 0
def score(self, x, y):
return np.mean(self.predict(x) == y)
def update_val_loss(self, x_val, y_val):
if x_val is None:
return
val_loss = 0
for i in range(len(x_val)):
z = self.forpass(x_val[i])
a = self.activation(z)
a = np.clip(a, 1e-10, 1-1e-10)
val_loss += -(y_val[i]*np.log(a) + (1 - y_val[i])*np.log(1-a))
self.val_losses.append(val_loss/len(y_val))
def __init__(self, learning_rate=0.1):
self.w = None
self.b = None
self.losses = []
self.val_losses = []
self.w_history = []
self.lr = learning_rate훈련 세트의 손실을 기록한 것 처럼 검증 세트에 대한 손실도 기록하도록 val_losses 변수를 추가합니다.
def fit(self, x, y, epochs=100, x_val=None, y_val=None):
self.w = np.ones(x.shape[1])
self.b = 0
self.w_history.append(self.w.copy())
np.random.seed(42)
for _ in range(epochs):
loss = 0
indexes = np.random.permutation(np.arange(len(x)))
for i in indexes:
z = self.forpass(x[i])
a = self.activation(z)
err = y[i] - a
w_grad, b_grad = self.backprop(x[i], err)
self.w -= self.lr*w_grad
self.b -= b_grad
self.w_history.append(self.w.copy())
a = np.clip(a, 1e-10, 1-1e-10)
loss += -(y[i]*np.log(a) + (1 - y[i])*np.log(1 - a))
self.losses.append(loss/len(y))
self.update_val_loss(x_val, y_val) fit()메서드에 검증 세트를 받을 수 있도록 x_val, y_val 매개변수를 추가하고, 검증 세트 손실을 계산하는 함수도 추가합니다.
def update_val_loss(self, x_val, y_val):
if x_val is None:
return
val_loss = 0
for i in range(len(x_val)):
z = self.forpass(x_val[i])
a = self.activation(z)
a = np.clip(a, 1e-10, 1-1e-10)
val_loss += -(y_val[i]*np.log(a) + (1 - y_val[i])*np.log(1-a))
self.val_losses.append(val_loss/len(y_val))검증 손실을 계산하는 함수는 위와 같이 정의합니다.
이제 모델을 수정하였으므로, 전처리된 훈련 세트와 검증 세트를 이용하여 단일층 신경망을 훈련해봅니다.
layer3 = SingleLayer()
layer3.fit(x_train_scaled, y_train, x_val=x_val_scaled, y_val=y_val)
plt.ylim(0, 0.3)
plt.plot(layer3.losses)
plt.plot(layer3.val_losses)
plt.ylabel('loss')
plt.xlabel('epoch')
plt.legend(['train_loss', 'val_loss'])
plt.show()
위 그래프를 보면, 대략 20번째 이후로 검증 손실이 훈련 손실보다 커지는 것을 알 수 있습니다. 따라서 이 모델은 20번의 에포크 이후에는 훈련 할 필요가 없을 것 같으므로, 훈련을 조기 종료(early stopping)해도 될 것 같습니다.
layer4 = SingleLayer()
layer4.fit(x_train_scaled, y_train, epochs=20)
layer4.score(x_val_scaled, y_val)
규제 방법을 배우고 단일층 신경망에 적용합니다
이전에 과대적합을 해결하는 방법 중 하나로 가중치 규제(regularization)을 설명했습니다. 규제는 말 그대로 특정 가중치가 너무 커지지 않게 규제하는 기법입니다. 가중치를 규제하면 일반화 성능이 올라갑니다.
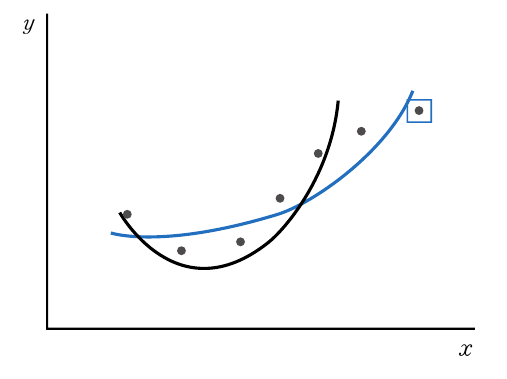
위 그림을 보면, 두 그래프 모두 어느정도 데이터를 잘 나타내고 있습니다. 이 경우, 경사가 급한 그래프보다 완만한 그래프가 박스 표시된 점을 더 잘 표현하기 때문에, 완만한 그래프가 더 좋다고 평가합니다. 그렇다면 만약 모든 점을 완벽하게 표현하는 그래프는 어떨까요?

위 그래프는 기존의 6개의 점을 완벽하게 표현하지만, 박스 표시된 새로운 점은 표현하지 못합니다. 모델이 기존의 데이터에만 집착하여 훈련되었기 때문에 새로운 데이터에 적응하지 못하므로, 좋은 성능을 가졌다고 할 수 없습니다. 이런 경우를 '모델이 일반화되지 않았다'라고 합니다. 이럴 때 규제를 사용하여 가중치를 제한하면 모델이 몇 개의 데이터에 집착하는 것을 막아 일반화 성능을 높일 수 있습니다.
예를 들어, 어떤 모델이 가중치가 6개인 6차방정식 형태로 과적합 되었을 때, 규제를 사용하면 이 중 몇 개의 가중치를 제한하거나 아예 없애버려서 2차, 3차 형태로 낮춰 좀 더 완만한 그래프를 가질 수 있게 해주기도 합니다.
L1 규제
L1 규제는 손실 함수에 가중치의 절댓값인 L1 norm을 추가합니다. L1 norm은 다음과 같이 정의됩니다.
$||\pmb{w}||_1 = \sum_{i=1}^n|w_i|$
선형대수에서 볼드체 소문자는 벡터, 대문자는 행렬을 의미합니다.
여기에서 L1 norm의 $n$은 가중치의 개수입니다. 따라서 L1 규제는 '가중치의 절댓값을 손실 함수에 더한 것'이라고 생각해도 됩니다. 이제 로지스틱 함수에 L1 규제를 적용해보겠습니다. 이 때, 가중치의 절댓값을 손실함수에 그냥 더하는 것이 아닌, 규제의 크기를 조절하는 파라미터를 곱하여 더합니다.
$L = -(y\log(a) + (1-y)\log(1-a))+\alpha\sum_{i=1}^n|w_i|$
$\alpha$는 규제의 양을 조절하는 하이퍼파라미터입니다. $\alpha$의 값이 크면 전체 손실 함수의 값이 커지므로, $\pmb{w}$의 합이 작아져야 합니다. 즉, 가중치의 크기가 줄어듭니다. 반대로 $\alpha$값이 작으면 $\pmb{w}$의 합이 커져도 손실 함수가 큰 폭으로 커지지 않습니다. 이제 경사 하강법을 적용하기 위해 규제를 적용한 위의 손실 함수를 미분해보겠습니다.
L1 규제의 미분
이전에 로지스틱 손실 함수를 미분해보았으므로, L1 규제만 미분해서 더하면 됩니다. $|\pmb{w}|$를 $\pmb{w}$에 대해 미분하면 부호만 남습니다. 따라서 손실 함수의 미분은 다음과 같습니다.
$\frac{\partial}{\partial \pmb{w}}L = -(y-a)x + \alpha\times sign(\pmb{w})$
가중치를 업데이트 할 때는 학습률을 곱하여 기울기를 뺐으므로, 가중치 업데이트 식은 다음과 같습니다.
$\pmb{w} = \pmb{w} -\eta\frac{\partial}{\partial \pmb{w}}L =
\pmb{w} +\eta((y-a)x - \alpha\times sign(\pmb{w}))$
로지스틱 손실 함수는 이진 분류 모델에서 사용하는 손실 함수였습니다. 분류 모델 말고도 회귀 모델에도 같은 원리로 손실 함수, 제곱 오차에 규제를 적용할 수 있는데, 이를 라쏘(Lasso) 모델이라고 합니다. 라쏘는 특정 가중치를 0으로 만들어버릴 정도로 규제를 할 수도 있습니다. 가중치가 0인 특성은 모델에서 사용하지 않는다는 의미이므로 모델의 복잡도를 줄이는 효과를 줍니다.
미분 결과에서 알 수 있듯이, L1 규제는 하이퍼파라미터 $\alpha$에 많이 의존합니다. 즉, 가중치의 크기에 따라 규제의 크기가 변하지 않으므로 규제 효과가 좋다고 말하기 어렵습니다.
L2 규제
L2 규제는 손실 함수에 가중치에 대한 L2 norm의 제곱을 더합니다. L2 norm은 다음과 같이 정의됩니다.
$||\pmb{w}||_2 = \sqrt{\sum_{i=1}^n|w_i|^2}$
손실 함수에 L2 norm의 제곱을 더하면 L2 규제가 됩니다. L2 norm을 제곱하면 제곱근이 없어지면서 2차식 형태가 나옵니다. 따라서 미분의 결과를 깔끔하게 만들기 위해 손실함수에 L2 norm의 제곱을 더하면서 계수로 $\frac{1}{2}$을 곱해줍니다.
$L = -(y\log(a) + (1-y)\log(1-a))+\frac{1}{2}\alpha\sum_{i=1}^n|w_i|^2$
L2 규제의 미분
L1규제를 미분할 때와 같은 방법으로 미분합니다.
$\frac{\partial}{\partial \pmb{w}}L = -(y-a)x + \alpha\times \pmb{w}$
이 결과를 가중치 업데이트 식에 적용하면 다음과 같습니다.
$\pmb{w} = \pmb{w} -\eta\frac{\partial}{\partial \pmb{w}}L =
\pmb{w} +\eta((y-a)x - \alpha\times \pmb{w})$
L2 규제는 그래이디언트 계산에 가중치의 값이 포함되므로 가중치의 부호만 사용하는 L1규제보다는 좀 더 효과적이며, 가중치를 완전히 0으로 만들지는 않습니다.
회귀 모델에 L2 규제를 적용한 것을 릿지(Ridge)모델이라고 합니다.
로지스틱 회귀에 규제 적용해보기
이전에 작성했던 SingleLayer클래스를 수정하여 규제를 추가해보겠습니다. 실전에서는 주로 L2 규제를 사용하나, 차이점을 알아보기 위해 두 규제 모두 구현해봅니다.
전체 코드는 다음과 같습니다.
class SingleLayer:
def __init__(self, learning_rate=0.1, l1=0, l2=0):
self.w = None
self.b = None
self.losses = []
self.val_losses = []
self.w_history = []
self.lr = learning_rate
self.l1 = l1
self.l2 = l2
def forpass(self, x):
z = np.sum(x*self.w) + self.b
return z
def backprop(self, x, err):
w_grad = -err*x
b_grad = -err*1
return w_grad, b_grad
def activation(self, z):
a = 1 / (1 + np.exp(-z))
return a
def fit(self, x, y, epochs=100, x_val=None, y_val=None):
self.w = np.ones(x.shape[1])
self.b = 0
self.w_history.append(self.w.copy())
np.random.seed(42)
for _ in range(epochs):
loss = 0
indexes = np.random.permutation(np.arange(len(x)))
for i in indexes:
z = self.forpass(x[i])
a = self.activation(z)
err = y[i] - a
w_grad, b_grad = self.backprop(x[i], err)
w_grad += self.l1*np.sign(self.w) + self.l2*self.w
self.w -= self.lr*w_grad
self.b -= b_grad
self.w_history.append(self.w.copy())
a = np.clip(a, 1e-10, 1-1e-10)
loss += -(y[i]*np.log(a) + (1 - y[i])*np.log(1 - a))
self.losses.append(loss/len(y) + self.reg_loss())
self.update_val_loss(x_val, y_val)
def predict(self, x):
z = [self.forpass(x_i) for x_i in x]
return np.array(z) > 0
def score(self, x, y):
return np.mean(self.predict(x) == y)
def update_val_loss(self, x_val, y_val):
if x_val is None:
return
val_loss = 0
for i in range(len(x_val)):
z = self.forpass(x_val[i])
a = self.activation(z)
a = np.clip(a, 1e-10, 1-1e-10)
val_loss += -(y_val[i]*np.log(a) + (1 - y_val[i])*np.log(1-a))
self.val_losses.append(val_loss/len(y_val) + self.reg_loss())
def reg_loss(self):
return self.l1*np.sum(np.abs(self.w)) + self.l2 / 2*np.sum(self.w**2)
class SingleLayer:
def __init__(self, learning_rate=0.1, l1=0, l2=0):
self.w = None
self.b = None
self.losses = []
self.val_losses = []
self.w_history = []
self.lr = learning_rate
self.l1 = l1
self.l2 = l2__init__() 메서드에 L1과 L2 규제의 강도를 조절하는 매개변수를 추가합니다. 나중에 가중치 계산 시 L1과 L2규제를 둘 다 한번에 적용할 것인데, 기본값은 0이므로, 클래스 생성 시 해당 규제의 값을 설정해주지 않으면 그 규제는 사용하지 않습니다.
def reg_loss(self):
return self.l1*np.sum(np.abs(self.w)) + self.l2 / 2*np.sum(self.w**2)새로운 메서드를 추가해줍니다. 손실 함수에 규제를 더하는 페널티 항이 추가되어야 합니다. 위에서 언급했듯이, L1과 L2 한번에 더합니다.
def fit(self, x, y, epochs=100, x_val=None, y_val=None):
self.w = np.ones(x.shape[1])
self.b = 0
self.w_history.append(self.w.copy())
np.random.seed(42)
for _ in range(epochs):
loss = 0
indexes = np.random.permutation(np.arange(len(x)))
for i in indexes:
z = self.forpass(x[i])
a = self.activation(z)
err = y[i] - a
w_grad, b_grad = self.backprop(x[i], err)
w_grad += self.l1*np.sign(self.w) + self.l2*self.w
self.w -= self.lr*w_grad
self.b -= b_grad
self.w_history.append(self.w.copy())
a = np.clip(a, 1e-10, 1-1e-10)
loss += -(y[i]*np.log(a) + (1 - y[i])*np.log(1 - a))
self.losses.append(loss/len(y) + self.reg_loss())
self.update_val_loss(x_val, y_val)fit() 메서드에서 역방향 계산 수행 시 그래이디언트에 규제의 미분값을 더합니다. 이전에도 말했듯이, 가중치 업데이트에 한번에 두 규제를 모두 더합니다.
def update_val_loss(self, x_val, y_val):
if x_val is None:
return
val_loss = 0
for i in range(len(x_val)):
z = self.forpass(x_val[i])
a = self.activation(z)
a = np.clip(a, 1e-10, 1-1e-10)
val_loss += -(y_val[i]*np.log(a) + (1 - y_val[i])*np.log(1-a))
self.val_losses.append(val_loss/len(y_val) + self.reg_loss())마지막으로, 검증 세트의 손실 계산에도 페널티 항을 추가해줍니다.
이제 위 모델을 훈련해보겠습니다. 먼저, L1 규제를 적용한 모델입니다.
l1_list = [0.0001, 0.001, 0.01]
for l1 in l1_list:
lyr = SingleLayer(l1=l1)
lyr.fit(x_train_scaled, y_train, x_val=x_val_scaled, y_val=y_val)
plt.plot(lyr.losses)
plt.plot(lyr.val_losses)
plt.title('Learning Curve (l1={})'.format(l1))
plt.ylabel('loss')
plt.xlabel('epoch')
plt.legend(['train_loss', 'val_loss'])
plt.ylim(0, 0.3)
plt.show()
plt.plot(lyr.w, 'bo')
plt.title('Weight (l1={})'.format(l1))
plt.ylabel('value')
plt.xlabel('weight')
plt.ylim(-4, 4)
plt.show()



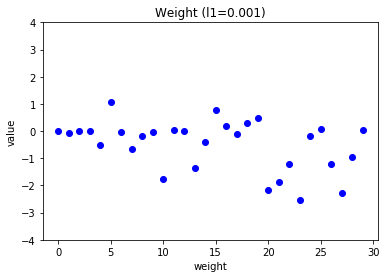


위 그래프를 보면 규제가 커질수록 가중치들은 작아지고, 훈련 세트와 검증 세트의 손실이 모두 커지는 것을 볼 수 있습니다. 즉, 과소적합 현상이 보입니다. 위 그래프들을 보니 적절한 규제 하이퍼파라미터 값은 0.001같습니다.
모델의 성능을 한번 확인해보겠습니다.
layer5 = SingleLayer(l1=0.001)
layer5.fit(x_train_scaled, y_train, epochs=20)
layer5.score(x_val_scaled, y_val)
규제를 적용하기 전과 결과가 같은데, 이는 데이터 세트가 작기 때문입니다.
이제 L2 규제를 적용해보겠습니다.
l2_list = [0.0001, 0.001, 0.01]
for l2 in l2_list:
lyr = SingleLayer(l2=l2)
lyr.fit(x_train_scaled, y_train, x_val=x_val_scaled, y_val=y_val)
plt.plot(lyr.losses)
plt.plot(lyr.val_losses)
plt.title('Learning Curve (l2={})'.format(l2))
plt.ylabel('loss')
plt.xlabel('epoch')
plt.legend(['train_loss', 'val_loss'])
plt.ylim(0, 0.3)
plt.show()
plt.plot(lyr.w, 'bo')
plt.title('Weight (l2={})'.format(l2))
plt.ylabel('value')
plt.xlabel('weight')
plt.ylim(-4, 4)
plt.show()





사이킷런을 사용하여 규제 적용해보기
sgd = SGDClassifier(loss='log', penalty='l2', alpha=0.001, random_state=42)
sgd.fit(x_train_scaled, y_train)
sgd.score(x_val_scaled, y_val)
교차 검증
교차 검증은 전체 데이터 세트의 샘플이 충분하지 않을 때, 검증 세트를 훈련 세트에서 분리하느라 훈련 세트의 데이터가 부족해질 때 사용하면 좋습니다.

교차 검증은 훈련 세트를 위와 같이 작은 덩어리로 나눕니다. 이 때, 나눠진 각각의 훈련세트를 '폴드'라고 합니다.
교차 검증의 과정은 다음과 같습니다.
1. 훈련 세트를 k개의 폴드(fold)로 나눕니다.
2. 첫 번째 폴드를 검증 세트로 사용하고, 나머지 폴드는 훈련 세트로 사용합니다.
3. 모델을 훈련하고, 검증 세트로 평가합니다.
4. 차례대로 다음 폴드를 검증 세트로 사용하며, 2~3을 반복합니다.
5. k개의 검증 세트로 k번 성능을 평가한 후, 평균을 내서 최종 성능을 계산합니다.
교차 검증은 훈련 세트를 k개의 폴드로 나누므로 k-폴드 교차 검증이라고 합니다.
k-폴드 교차 검증 구현해보기
validation_scores=[]
k = 10
bins = len(x_train_all) // k
for i in range(k):
start = i*bins
end = (i+1)*bins
val_fold = x_train_all[start:end]
val_target = y_train_all[start:end]
train_index = list(range(0, start)) + list(range(end, len(x_train)))
train_fold = x_train_all[train_index]
train_target = y_train_all[train_index]
train_mean = np.mean(train_fold, axis=0)
train_std = np.std(train_fold, axis=0)
train_fold_scaled = (train_fold - train_mean) / train_std
val_fold_scaled = (val_fold - train_mean) / train_std
lyr = SingleLayer(l2=0.01)
lyr.fit(train_fold_scaled, train_target, epochs=50)
score=lyr.score(val_fold_scaled, val_target)
validation_scores.append(score)
print(np.mean(validation_scores))
위 코드에서 중요한 점은 훈련 데이터의 전처리를 폴드를 나눈 후에 수행한다는 점입니다. 만약 폴드를 나누기 전에 전처리를 한다면 이는 검증 폴드의 정보를 누설하게 되는 것입니다.
사이킷런에서 교차검증 구현하기
from sklearn.model_selection import cross_validate
sgd = SGDClassifier(loss='log', penalty='l2', alpha=0.001, random_state=42)
scores = cross_validate(sgd, x_train_all, y_train_all, cv=10)
print(np.mean(scores['test_score']))
정확도는 85%로 생각보다 낮은데, 이는 전처리를 하지 않았기 때문입니다.
사이킷런에서 전처리 단계를 포함하여 교차 검증 구현하기
앞에서 교차 검증을 구현할 때는 폴드를 나누고 나서, 검증 폴드를 전처리했습니다. 그런데 지금 위의 사이킷런 코드에서는 이렇게 폴드를 나누고 나서 전처리를 할만한 부분이 없습니다. 그렇다고 훈련 데이터를 모두 전처리 해버리고 cross_validate() 의 입력으로 넣어버리면 검증 폴드가 누설되어버립니다.
사이킷런은 검증 폴드가 전처리 단계에서 누설되지 않게 전처리 단계와 모델 클래스를 이어주는 Pipeline이라는 클래스를 제공합니다.
from sklearn.pipeline import make_pipeline
from sklearn.preprocessing import StandardScaler
pipe = make_pipeline(StandardScaler(), sgd)
scores = cross_validate(pipe, x_train_all, y_train_all, cv=10, return_train_score=True)
print(np.mean(scores['train_score']))
print(np.mean(scores['test_score']))
make_pipeline()함수로 pipeline 객체를 만들고, 이를 cross_validate() 함수에 전달합니다. 이러면 cross_validate 함수는 훈련 세트를 훈련 폴드와 검증 폴드로 나누기만 하고, 전처리 단계는 매개변수로 전달된 pipeline 객체에서 이루어집니다.
'Study > MachineLearning' 카테고리의 다른 글
| [Do it!] 6. 이미지 분류하기 - 합성곱 신경망 (0) | 2021.09.07 |
|---|---|
| [Do it!] 5. 다중 분류 (0) | 2021.09.06 |
| [Do it!] 4. 2개의 층 연결하기 - 다층 신경망 (0) | 2021.09.06 |
| [Do it!] 2. 분류하는 뉴런 만들기 - 이진 분류 (0) | 2021.09.03 |
| [Do it!] 1. 머신러닝 기초 - 수치 예측 (0) | 2021.09.02 |